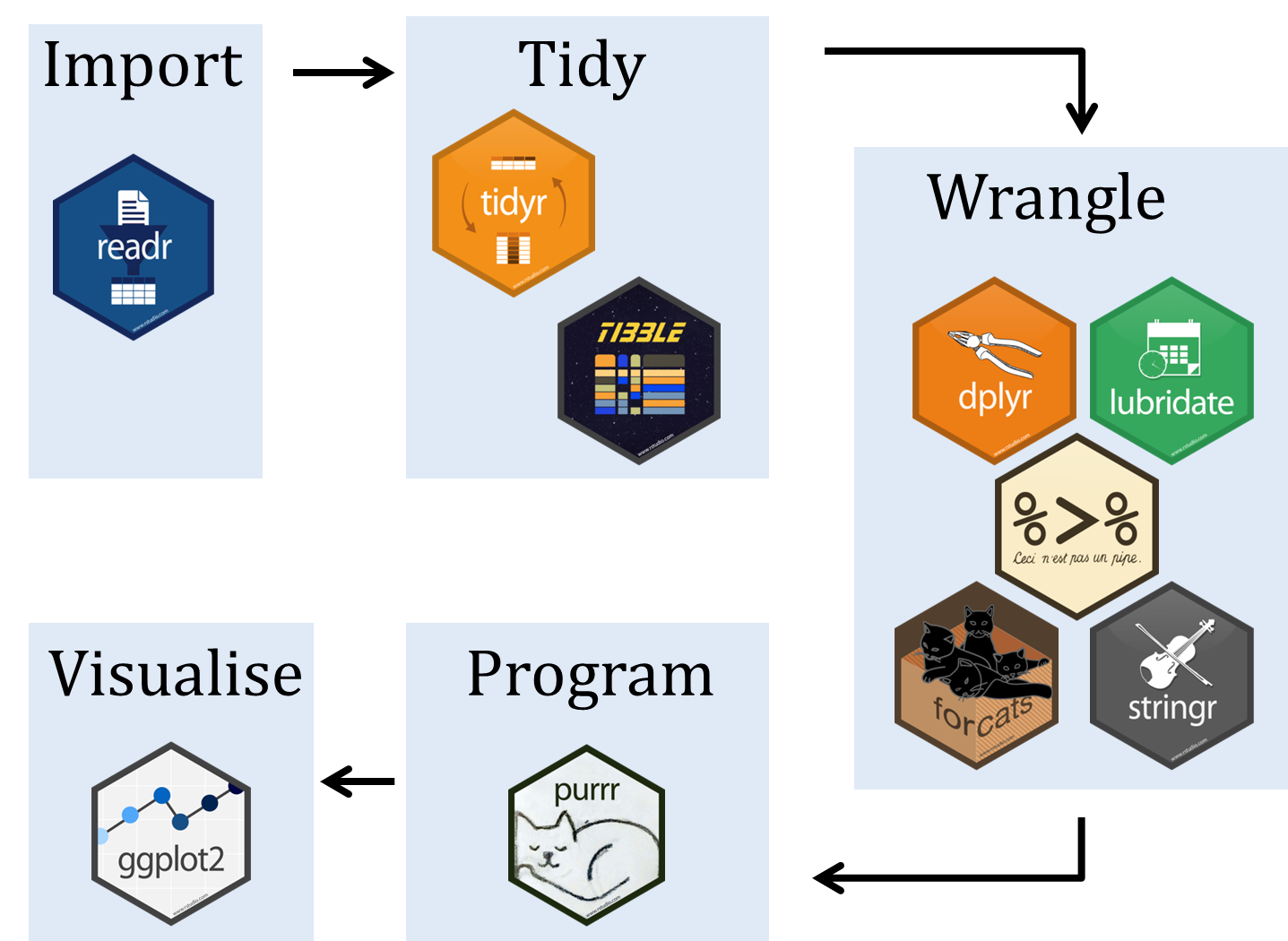
Agenda
Introdução ao R
- Tidyverse

- Tidyverse
Introdução ao Python
- Pandas

- Pandas
Visualização de dados

Introdução ao R
História do R
R é uma linguagem de programação de alto nível com ambiente voltado para visualização e análise de dados. Em essência foi inspirada na linguagem de programação S. Foi iniciamente escrita por Ross Ihaka e Robert Gentleman no departamento de estatística da universidade de Auckland.

Robert Gentleman

Ross Ihaka
Conceitos básicos
Conceitos básicos
Atribuimos valor usando uma seta <- apontada para a variável
Declaração de variáveis
numerico <- 123 double <- 123.2 inteiro <- 321L complexo <- 321i booleano <- TRUE caractere <- "Bem-vindos"Conceitos básicos
Atribuimos valor usando uma seta <- apontada para a variável
Declaração de variáveis
numerico <- 123 double <- 123.2 inteiro <- 321L complexo <- 321i booleano <- TRUE caractere <- "Bem-vindos"Acessando os valores
print(numerico)## [1] 123 caractere## [1] "Bem-vindos"Tipos de dados estruturados
Podemos declarar um vetor usando c() e uma lista usando list()
Vetores
vetor_int <- c(1, 3, 5) vetor_char <- c("teste", "teste", "teste") vetor_bool <- c(TRUE, TRUE, FALSE) vetor_int## [1] 1 3 5Listas
lista_int <- list(1, 3, 5) lista_lista <- list(1, 2.3, list("tres")) lista_lista## [[1]]## [1] 1## ## [[2]]## [1] 2.3## ## [[3]]## [[3]][[1]]## [1] "tres"Qual é a diferença?
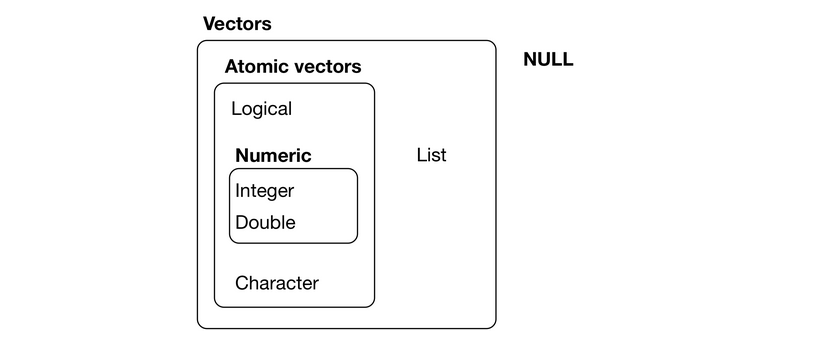
Vetores são atômicos, só aceitam um tipo:
vetor_diferente <- c(1, 2.5, TRUE, "ola") vetor_diferente## [1] "1" "2.5" "TRUE" "ola" is.atomic(vetor_diferente)## [1] TRUELista não são atômicas, aceitam diversos tipos:
lista_diferente <- list(1, TRUE, "ola") lista_diferente## [[1]]## [1] 1## ## [[2]]## [1] TRUE## ## [[3]]## [1] "ola" is.atomic(lista_diferente)## [1] FALSEHierarquia de tipo primitivos
O R possui uma conversão de tipos, sendo assim, garantindo que todo vetor seja atômico.
 R 4 Data Science - Hadley
R 4 Data Science - Hadley
Hierarquia de tipo primitivos
Os tipos mais fortes são:
- character
- complex
- numeric
- logical
Conversão:
v1 <- c(FALSE, "tipo", 5)Hierarquia de tipo primitivos
Os tipos mais fortes são:
- character
- complex
- numeric
- logical
Conversão:
v1 <- c(FALSE, "tipo", 5)v1## [1] "FALSE" "tipo" "5"Hierarquia de tipo primitivos
Os tipos mais fortes são:
- character
- complex
- numeric
- logical
Conversão:
v1 <- c(FALSE, "tipo", 5)v1## [1] "FALSE" "tipo" "5"v2 <- c(32, 1, 5i)Hierarquia de tipo primitivos
Os tipos mais fortes são:
- character
- complex
- numeric
- logical
Conversão:
v1 <- c(FALSE, "tipo", 5)v1## [1] "FALSE" "tipo" "5"v2 <- c(32, 1, 5i)v2## [1] 32+0i 1+0i 0+5iHierarquia de tipo primitivos
Os tipos mais fortes são:
- character
- complex
- numeric
- logical
Conversão:
v1 <- c(FALSE, "tipo", 5)v1## [1] "FALSE" "tipo" "5"v2 <- c(32, 1, 5i)v2## [1] 32+0i 1+0i 0+5iTipos:
typeof(v1)## [1] "character"typeof(v2)## [1] "complex"Data Frame
Data Frame
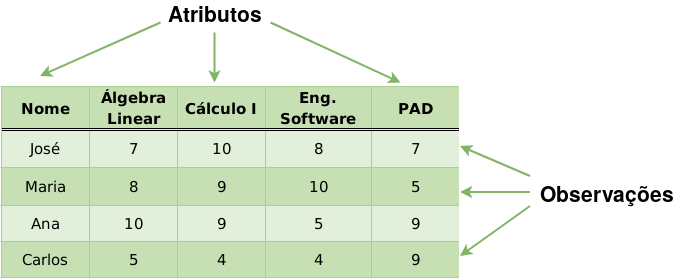
Matriz composta por linhas e colunas, cujas colunas representam as variáveis (atributos) e as linhas representam observações
Data Frame
Matriz composta por linhas e colunas, cujas colunas representam as variáveis (atributos) e as linhas representam observações
Podemos criar um dataframe usando data.frame()
meu_df <- data.frame( nome=c("Ana", "João"), idade=c(23, 24), cargo=c("Cientista", "Analista") ) meu_df## nome idade cargo## 1 Ana 23 Cientista## 2 João 24 AnalistaAcessando os atributos
meu_df$nome## [1] Ana João## Levels: Ana JoãoTipo
typeof(meu_df)## [1] "list"Classe
class(meu_df)## [1] "data.frame"Estrutura de decisão
O if do R é bem parecido com o do Java
valor_a <- 21 valor_b <- 42 if(valor_a < valor_b){ print("Valor A menor do que o valor B") } else if(valor_a == valor_b){ print("Valor A é igual ao valor B") } else { print("Valor A é maior do que o valor B") }## [1] "Valor A menor do que o valor B"Estrutura de decisão
O if do R é bem parecido com o do Java
valor_a <- 21 valor_b <- 42 if(valor_a < valor_b){ print("Valor A menor do que o valor B") } else if(valor_a == valor_b){ print("Valor A é igual ao valor B") } else { print("Valor A é maior do que o valor B") }## [1] "Valor A menor do que o valor B"Dica
ifelse(42 > TRUE, "Verdade universal", "Fake news")## [1] "Verdade universal"Estrutura de repetição
Por outro lado, o for do R parece com a sintaxe do Python
meu_vetor <- c(1, 2, 3) for(i in meu_vetor){ print(i) }## [1] 1## [1] 2## [1] 3Tidyverse 
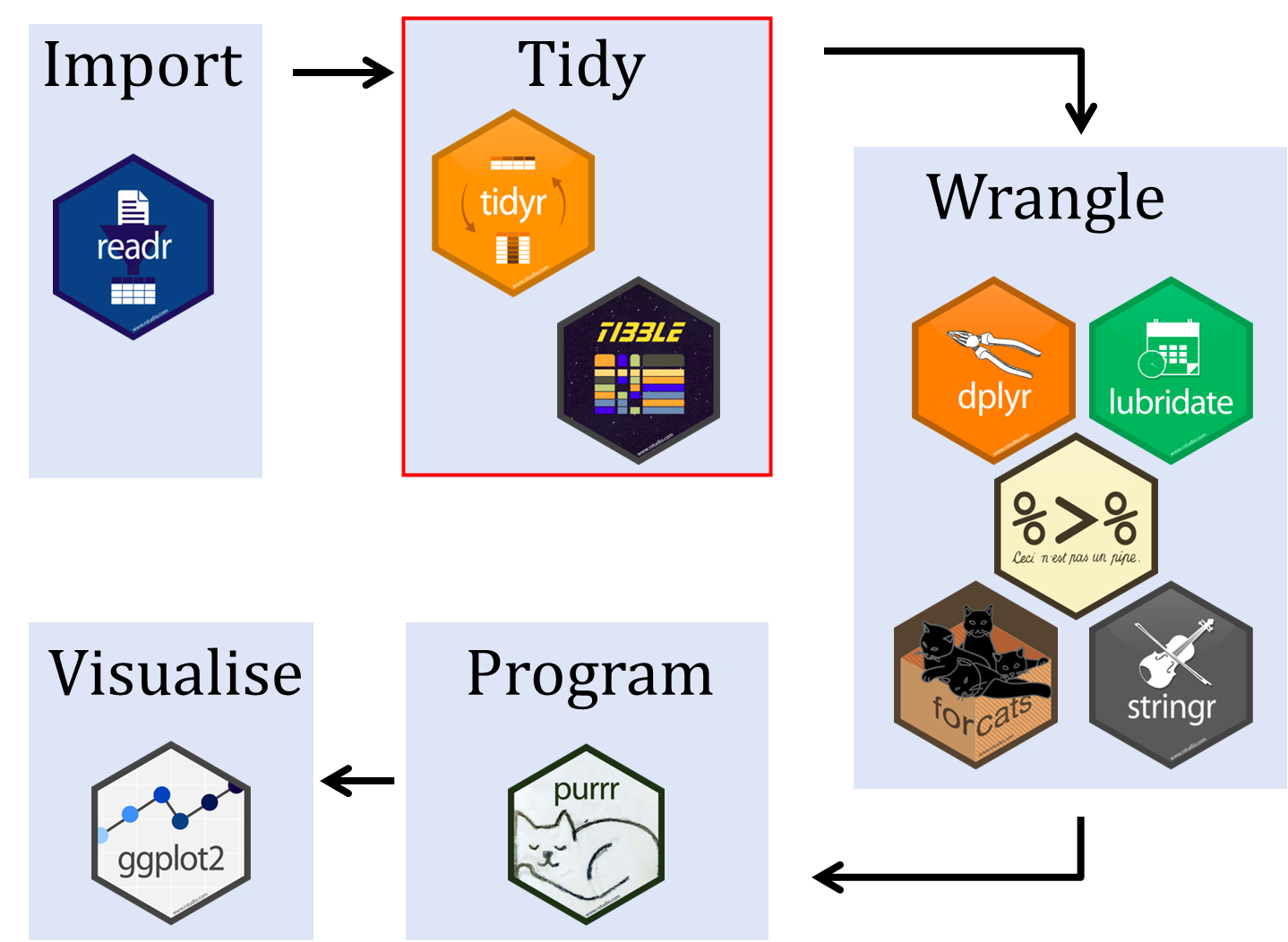
Tidyverse
Conjunto de pacotes em R para ciência de dados
Tidyverse
Conjunto de pacotes em R para ciência de dados
Processo de ciência de dados
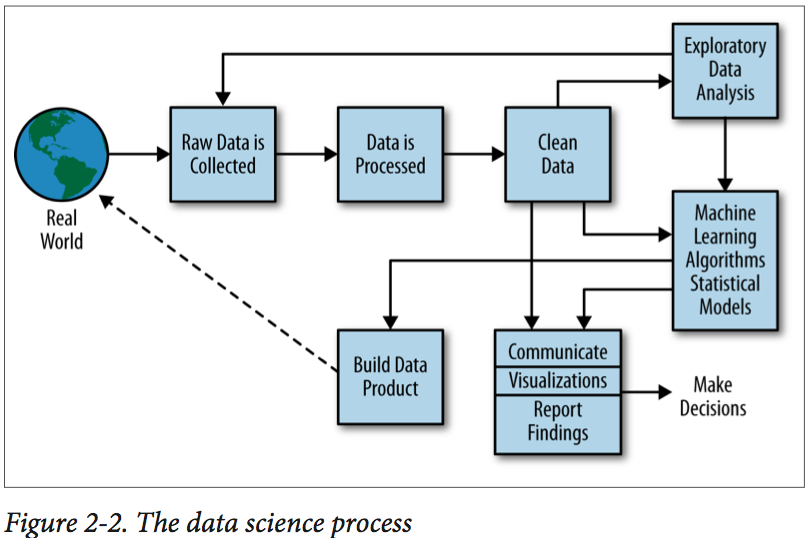
Doing Data Science - Rachel Schutt
Tidyverse
Conjunto de pacotes em R para ciência de dados
Processo de ciência de dados
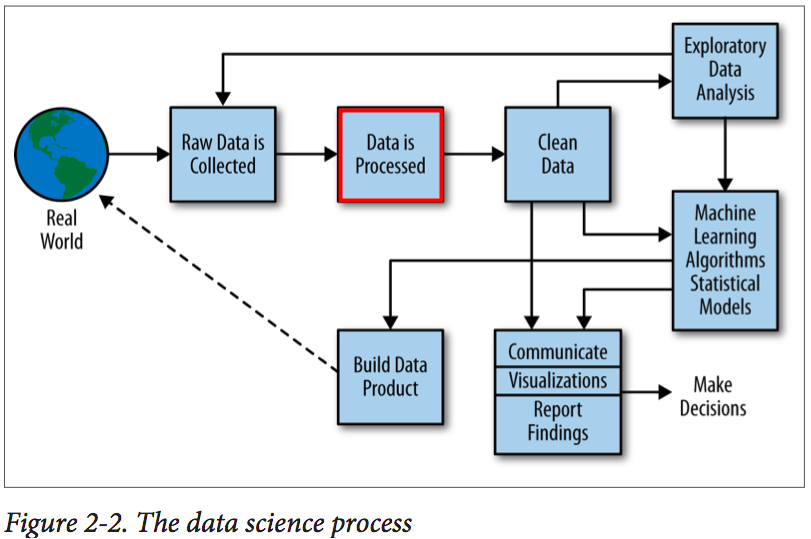
Doing Data Science - Rachel Schutt
Tidyverse
Conjunto de pacotes em R para ciência de dados
Processo de ciência de dados
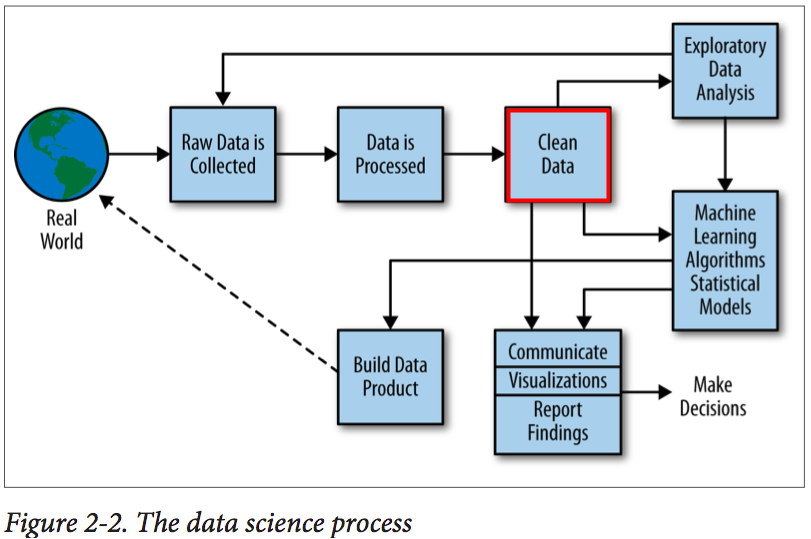
Doing Data Science - Rachel Schutt
Tidyverse
Conjunto de pacotes em R para ciência de dados
Processo de ciência de dados
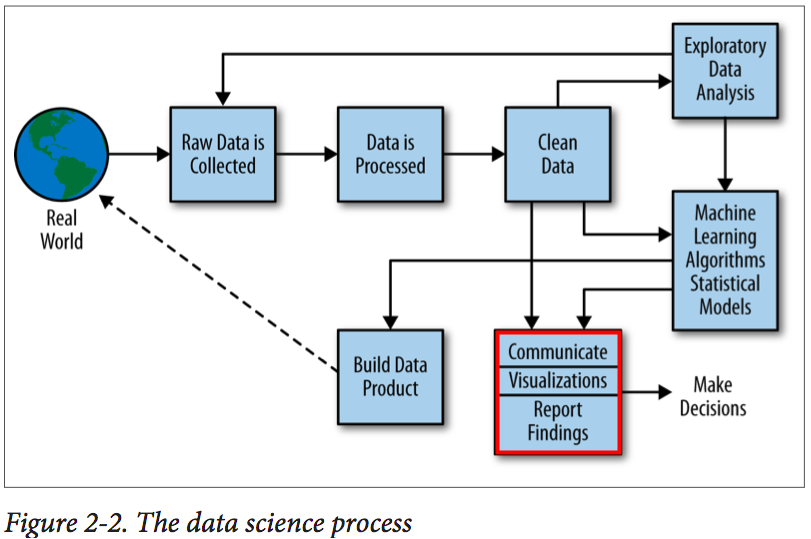
Doing Data Science - Rachel Schutt
Leitura e escrita de dados - readr
Leitura
library(readr) # Leitura dos dados star_wars <- readr::read_csv(file = "data/starwars.csv")## Parsed with column specification:## cols(## X1 = col_double(),## name = col_character(),## height = col_double(),## mass = col_double(),## hair_color = col_character(),## skin_color = col_character(),## eye_color = col_character(),## birth_year = col_double(),## gender = col_character(),## homeworld = col_character(),## species = col_character()## )Escrita
# escrita readr::write_csv(x = star_wars, path = "data/dado.csv")Manipulação de dados - dplyr
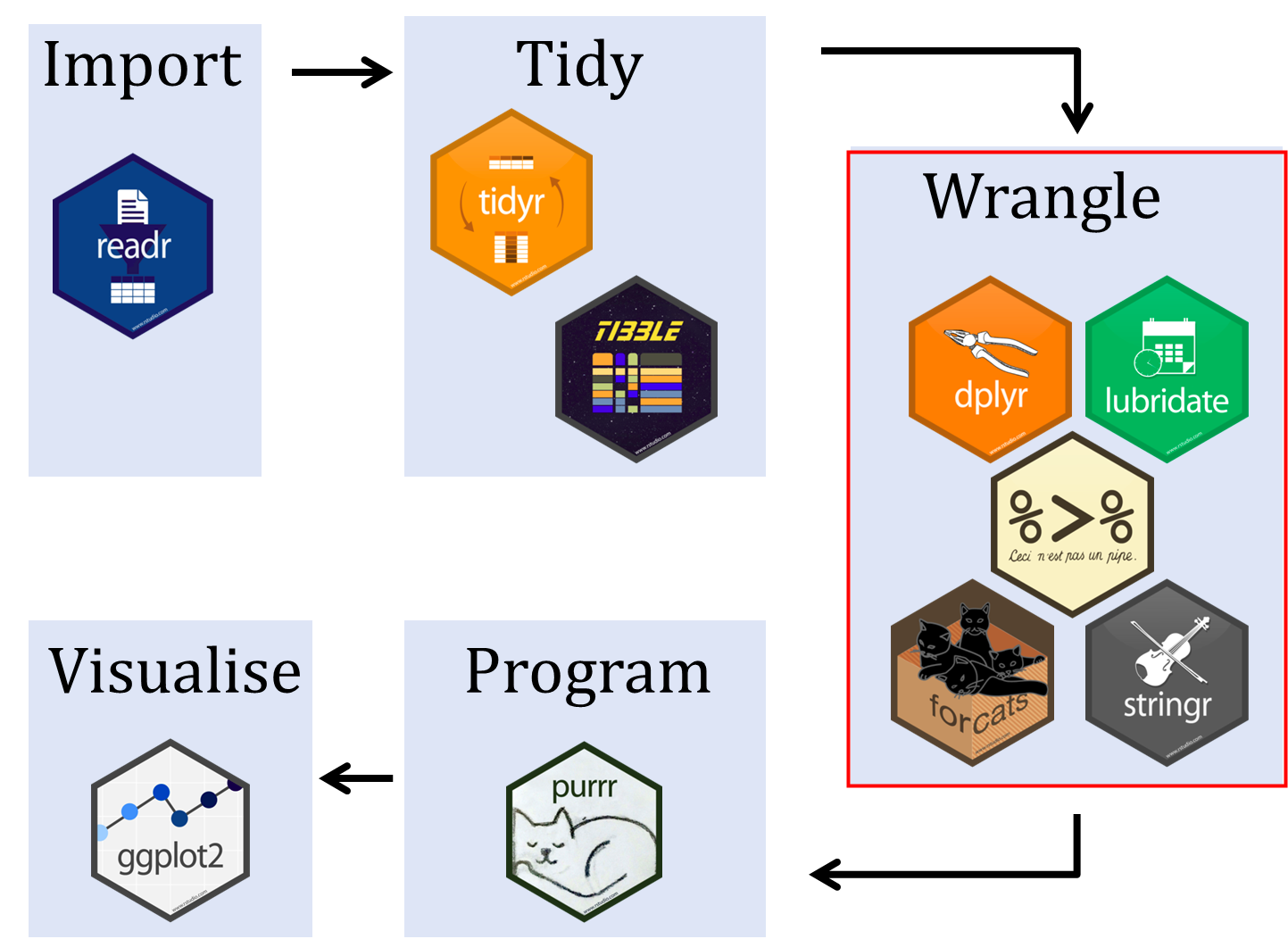
Para manipular nosso dataframe, vamos usar o pacote dplyr. Métodos básicos:

R for Data Science - Garrett Grolemund
Seleção e Filtro
Para facilitar a seleção dos atributos e tirar $, vamos usar o select():
library(dplyr) selecao <- dplyr::select(star_wars, name, hair_color) head(selecao, 3)## # A tibble: 3 x 2## name hair_color## <chr> <chr> ## 1 Luke Skywalker blond ## 2 C-3PO <NA> ## 3 R2-D2 <NA>Para filtrar por um valor específico, usamos filter():
filtro <- dplyr::filter(star_wars, species == "Droid" & skin_color == "gold") filtro## # A tibble: 1 x 11## X1 name height mass hair_color skin_color eye_color birth_year gender## <dbl> <chr> <dbl> <dbl> <chr> <chr> <chr> <dbl> <chr> ## 1 2 C-3PO 167 75 <NA> gold yellow 112 <NA> ## # … with 2 more variables: homeworld <chr>, species <chr>Agrupamento e agregação
Para criar grupos usamos a função group_by()
agrupamento <- dplyr::group_by(star_wars, species) head(agrupamento, 3)## # A tibble: 3 x 11## # Groups: species [2]## X1 name height mass hair_color skin_color eye_color birth_year gender## <dbl> <chr> <dbl> <dbl> <chr> <chr> <chr> <dbl> <chr> ## 1 1 Luke… 172 77 blond fair blue 19 male ## 2 2 C-3PO 167 75 <NA> gold yellow 112 <NA> ## 3 3 R2-D2 96 32 <NA> white, bl… red 33 <NA> ## # … with 2 more variables: homeworld <chr>, species <chr>Para criarmos uma agregação do nosso agrupamento, usamos summarise() e para ordenarmos arrange():
media_grupo <- dplyr::summarise(agrupamento, media = mean(height, na.rm = TRUE)) media_grupo_order <- dplyr::arrange(media_grupo, desc(media)) head(media_grupo_order, 3)## # A tibble: 3 x 2## species media## <chr> <dbl>## 1 Quermian 264## 2 Wookiee 231## 3 Kaminoan 221Transformação de atributos
Para criar/transformar novos atributos, usa-se mutate():
# conversão de cm para metros star_wars <- dplyr::mutate(star_wars, height = height/100) head(star_wars, 3)## # A tibble: 3 x 11## X1 name height mass hair_color skin_color eye_color birth_year gender## <dbl> <chr> <dbl> <dbl> <chr> <chr> <chr> <dbl> <chr> ## 1 1 Luke… 1.72 77 blond fair blue 19 male ## 2 2 C-3PO 1.67 75 <NA> gold yellow 112 <NA> ## 3 3 R2-D2 0.96 32 <NA> white, bl… red 33 <NA> ## # … with 2 more variables: homeworld <chr>, species <chr>Aplicando os métodos
Seleção
selecao <- dplyr::select(star_wars, -hair_color, -mass)Filtro
filtro <- dplyr::filter(selecao, eye_color == "blue")Agrupamento
agrupamento <- dplyr::group_by(filtro, species)Agregação
media_grupo <- dplyr::summarise(agrupamento, media = mean(height, na.rm = TRUE))Ordenação
media_grupo_order <- dplyr::arrange(media_grupo, desc(media))## # A tibble: 3 x 2## species media## <chr> <dbl>## 1 Wookiee 2.31## 2 Chagrian 1.96## 3 Tholothian 1.84Facilitando a vida com o operador pipe (%>%)
O pipe passa a resposta da primeira atribuição para o primeiro parâmetro da função seguinte:
library(magrittr) media_grupo <- star_wars %>% dplyr::select(-hair_color, -mass) %>% dplyr::filter(eye_color == "blue") %>% dplyr::group_by(species) %>% dplyr::summarise(media = mean(height, na.rm = TRUE)) %>% dplyr::arrange(desc(media)) head(media_grupo, 2)## # A tibble: 2 x 2## species media## <chr> <dbl>## 1 Wookiee 2.31## 2 Chagrian 1.96Facilitando a vida com o operador pipe (%>%)
Sem pipe
# Seleção selecao <- dplyr::select(star_wars, -hair_color, -mass) # Filtro filtro <- dplyr::filter(selecao, eye_color == "blue") # Agrupamento agrupamento <- dplyr::group_by(filtro, species) # Agregação media_grupo <- dplyr::summarise(agrupamento, media = mean(height, na.rm = TRUE)) # Ordenação media_grupo_order <- dplyr::arrange(media_grupo, desc(media))Com pipe
media_grupo <- star_wars %>% dplyr::select(-hair_color, -mass) %>% dplyr::filter(eye_color == "blue") %>% dplyr::group_by(species) %>% dplyr::summarise(media = mean(height, na.rm = TRUE)) %>% dplyr::arrange(desc(media))Exemplo de análise de dados
Exemplo de análise de dados
Dado de série temporal da temperatura da superfície terrestre
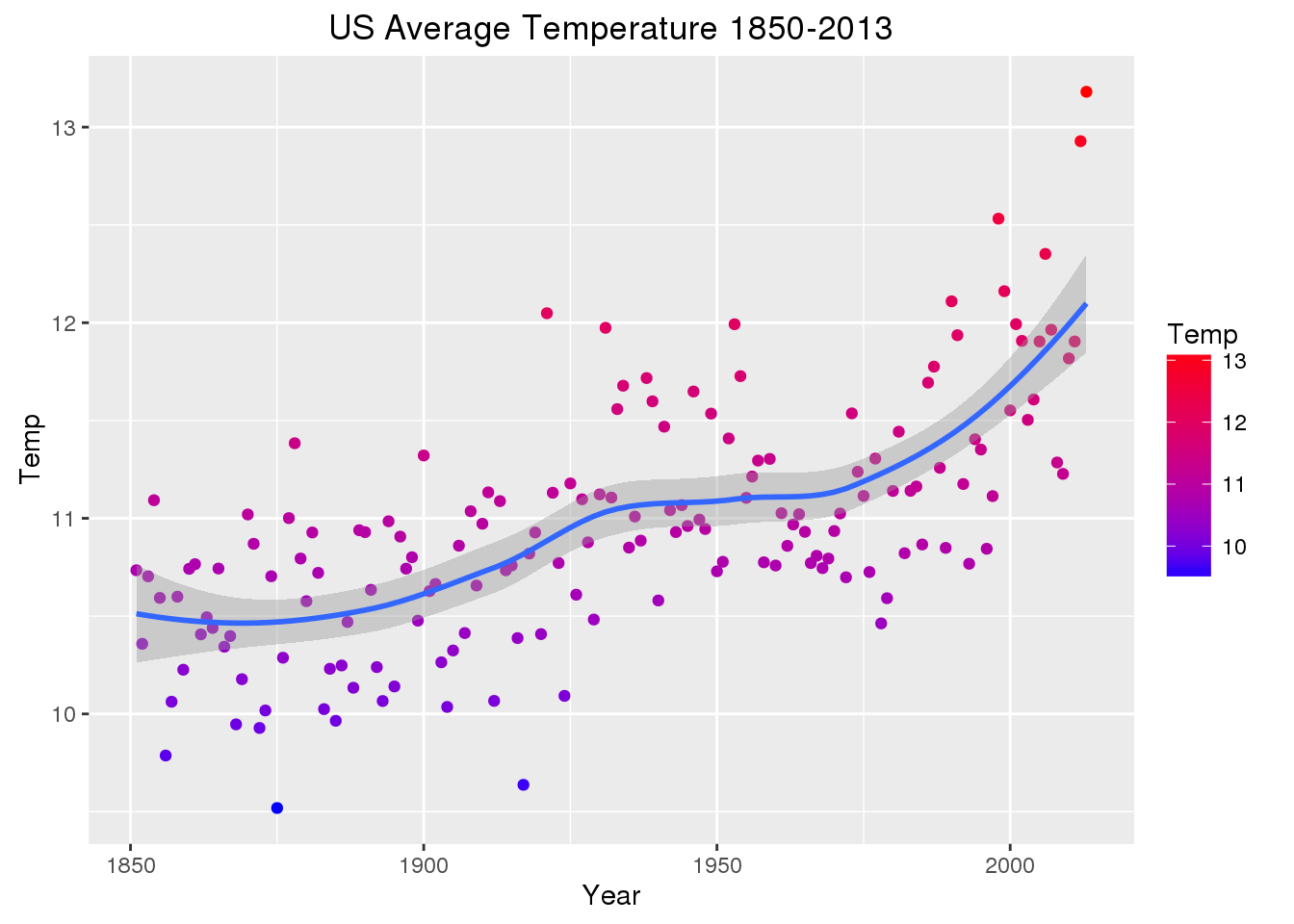 Fonte: Berkeley Earth
Fonte: Berkeley Earth
Exemplo de análise
# Leitura dos dados de mudança climática temperature_countries <- readr::read_csv("./data/GlobalLandTemperaturesByCountry.csv") # Leitura e seleção dos dados de continentes continent <- readr::read_csv("./data/countryContinent.csv") %>% dplyr::select(country, continent) # Seleção do atributo continente head(temperature_countries, 2)## # A tibble: 2 x 4## dt AverageTemperature AverageTemperatureUncertainty Country## <date> <dbl> <dbl> <chr> ## 1 1743-11-01 4.38 2.29 Åland ## 2 1743-12-01 NA NA Åland tail(temperature_countries, 2)## # A tibble: 2 x 4## dt AverageTemperature AverageTemperatureUncertainty Country ## <date> <dbl> <dbl> <chr> ## 1 2013-08-01 19.8 0.717 Zimbabwe## 2 2013-09-01 NA NA ZimbabweExemplo de análise
Filtro a partir do ano 2000 e extração da média anual
year_temperature <- temperature_countries %>% dplyr::filter(dt > "2000-01-01") %>% dplyr::mutate(dt = lubridate::year(dt)) %>% dplyr::group_by(Country, dt) %>% dplyr::summarise(year_mean = mean(AverageTemperature))## # A tibble: 3 x 3## # Groups: Country [1]## Country dt year_mean## <chr> <dbl> <dbl>## 1 Afghanistan 2000 16.7## 2 Afghanistan 2001 15.8## 3 Afghanistan 2002 15.5Junção dos continentes com cada país
continent_temperature <- year_temperature %>% dplyr::rename(country = Country) %>% dplyr::left_join(continent, by="country") %>% dplyr::filter(!is.na(continent))## # A tibble: 3 x 4## # Groups: country [1]## country dt year_mean continent## <chr> <dbl> <dbl> <chr> ## 1 Afghanistan 2000 16.7 Asia ## 2 Afghanistan 2001 15.8 Asia ## 3 Afghanistan 2002 15.5 AsiaExemplo de análise
Filtro a partir do ano 2000 e extração da média anual
year_temperature <- temperature_countries %>% dplyr::filter(dt > "2000-01-01") %>% dplyr::mutate(dt = lubridate::year(dt)) %>% dplyr::group_by(Country, dt) %>% dplyr::summarise(year_mean = mean(AverageTemperature))## # A tibble: 3 x 3## # Groups: Country [1]## Country dt year_mean## <chr> <dbl> <dbl>## 1 Afghanistan 2000 16.7## 2 Afghanistan 2001 15.8## 3 Afghanistan 2002 15.5Junção dos continentes com cada país
continent_temperature <- year_temperature %>% dplyr::rename(country = Country) %>% dplyr::left_join(continent, by="country") %>% dplyr::filter(!is.na(continent))## # A tibble: 3 x 4## # Groups: country [1]## country dt year_mean continent## <chr> <dbl> <dbl> <chr> ## 1 Afghanistan 2000 16.7 Asia ## 2 Afghanistan 2001 15.8 Asia ## 3 Afghanistan 2002 15.5 AsiaQual o continente que registrou a maior temperatura anual?
Qual o ano com a maior média de temperatura registrada?
Exemplo de análise
Qual o continente que registrou a maior temperatura anual?
continent_temperature %>% dplyr::group_by(continent) %>% summarise(maior_temp = max(year_mean, na.rm = TRUE)) ## # A tibble: 5 x 2## continent maior_temp## <chr> <dbl>## 1 Africa 30.3## 2 Americas 29.0## 3 Asia 29.7## 4 Europe 20.3## 5 Oceania 27.9Qual foi o ano com a maior média de temperatura registrada??
continent_temperature %>% dplyr::group_by(dt, continent) %>% dplyr::summarise(maior_temp = max(year_mean, na.rm = TRUE)) %>% dplyr::arrange(desc(maior_temp)) %>% head(5)## # A tibble: 5 x 3## # Groups: dt [5]## dt continent maior_temp## <dbl> <chr> <dbl>## 1 2000 Africa 30.3## 2 2010 Africa 30.1## 3 2012 Africa 29.9## 4 2009 Africa 29.9## 5 2011 Africa 29.8Introdução ao Python
Introdução ao Python
Python é uma linguagem multiparadigma, com uma sintaxe muito simples que permite ao programador focar no problema e deixar de lado questões da linguagem.
Comandos básicos da linguagem
Comandos básicos da linguagem
Declarações de variáveis
inteiro = 123 flutuante = 1.1234 booleano = True lista = [1, 2, 3, 4, 5] dicionario = {'chave': 'valor'} string = 'Um texto legal'Manipulação simples!
dicionario['chave'] ## 'valor' lista[0:3] ## [1, 2, 3] flutuante += 1 # 1.1234Estrutura de controle de decisão
As estruturas de controle de decisão facilitam o controle do fluxo que o código está tomando, representando parte importante da linguagem
if
if (2 > 1): print("Dois é maior que um!")## Dois é maior que um!else
if (2 < 1): print('Dois é menor que um')else: print('Dois é maior que um')## Dois é maior que umelif
if (1 > 1): print('Um é maior que um')elif(1 > 0.5): print('Um é maior que meio')## Um é maior que meioEstrutura de repetição
Essas são estruturas que permitem a criação de laços, havendo facilmente repetição de um certo bloco de código, e ainda, permite iterações em estruturas de dados como as listas
while
i = 0 while i < 5: print(i) i += 1 ## 0 ## 1 ## 2 ## 3 ## 4for-each
for i in [1, 2, 3]: print(i) ## 1 ## 2 ## 3 for i in range(0, 3): print(i) ## 0 ## 1 ## 2Pandas 🐼
Pandas 🐼
Pandas é uma biblioteca open source, com licença BSD, que fornece estruturas de dados de alto desempenho para a análise de dados na linguagem Python.
Para importar a biblioteca, utilize:
import pandas as pdEstruturas de dados básicas do Pandas
Estruturas de dados básicas do Pandas
Series
- Estruturas unidimensionais;
- Dados armazenados em linhas
- Índice nas linhas;
- Suporte a qualquer tipo de dados;
- Exemplo de aplicação: Séries Temporais
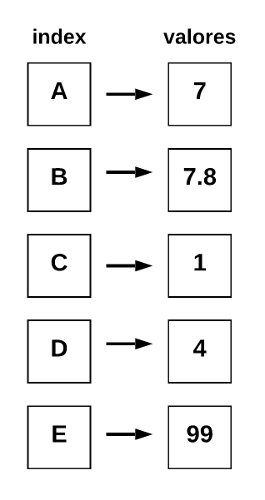
Estruturas de dados básicas do Pandas
Exemplos de utilização das Series
Criando uma Series simples
series = pd.Series([1, 2]) print(series) ## 0 1 ## 1 2 ## dtype: int64Series com índice
series = pd.Series([1, 2], [9, 'ultimo']) print(series) ## 9 1 ## ultimo 2 ## dtype: int64Recuperando valores
A recuperação dos dados é muito parecida com as encontradas em dicionários (chave-valor).
print(series[9]) ## 1 print(series["ultimo"]) ## 2 dicionario = {"Nome": "Maria"} print(dicionario["Nome"]) ## MariaEstruturas de dados básicas do Pandas
DataFrames
- Estruturas multidimensionais;
- Dados armazenados em linhas e colunas.
- Índice nas linhas e colunas;
- Suporte a qualquer tipo de dados (Um diferente para cada coluna).

Estruturas de dados básicas do Pandas
Exemplos de utilização dos DataFrames
Criando um DataFrame básico
df = pd.DataFrame([[1, 2, 3, 4], [11, 12, 13, 14]]) print(df) ## 0 1 2 3 ## 0 1 2 3 4 ## 1 11 12 13 14DataFrame com índice
df = pd.DataFrame([[1, 2, 3, 4], [11, 12, 13, 14]], index = ['um', 'dois']) print(df) ## 0 1 2 3 ## um 1 2 3 4 ## dois 11 12 13 14Índices e colunas
df = pd.DataFrame([[1, 2, 3, 4], [11, 12, 13, 14]], index = ['um', 'dois'], columns = ['a', 'b', 'c', 'd']) print(df) ## a b c d ## um 1 2 3 4 ## dois 11 12 13 14Recuperando valores
print(df['b']['um']) ## 2 print(df['a']['dois']) ## 11Manipulação básica de dados com Pandas
Manipulação básica de dados com Pandas
O pandas fornece uma infinidade de métodos para a seleção e filtro dos dados. Sendo todos úteis para o processo de análise de dados com Python
Manipulação básica de dados com Pandas
O pandas fornece uma infinidade de métodos para a seleção e filtro dos dados. Sendo todos úteis para o processo de análise de dados com Python
loc e iloc
Para iniciar, os métodos loc e iloc podem ser bastante utilizados para tais processos.
| DataFrame | Series |
|---|---|
| .loc[Nome da linha, Nome da coluna] | .loc[Nome da linha] |
| .iloc[posição da linha, posição da coluna] | .iloc[Posição da linha] |
Manipulação básica de dados com Pandas
Exemplo de utilização do loc e iloc
DataFrame de exemplo
df = pd.DataFrame({ 'nome': ['Joana', 'Maria', 'Josefa'], 'idade': [15, 18, 21], 'nota': [8, 9, 10] }, index = [7, 8, 9]) Recuperando a linha de nome 9 e a coluna de nome idade.
print(df.loc[9, 'idade']) ## 21É possível também recuperar mais de uma coluna ao mesmo tempo
print(df.loc[9, ['nome', 'idade']]) ## nome Josefa ## idade 21 ## Name: 9, dtype: object Para recuperar as posições, utilize o iloc.
print(df.iloc[2][['nome', 'idade']]) ## nome Josefa ## idade 21 ## Name: 9, dtype: objectFiltro de dados
Para muitas partes da análise de dados, realizar filtros e buscas é de extrema importância. No Pandas há diversos métodos que podem ser aplicados.
DataFrame de exemplo
df = pd.DataFrame({ 'nome': ['A1', 'A2', 'A3', 'A4'], 'valor': [5, 12, 8, 30] })filter
Filtra os dados pelo nome da coluna
df.filter(items = ['valor']) ## valor ## 0 5 ## 1 12 ## 2 8 ## 3 30
Filtrando os dados por uma coluna que contenha no nome a palavra lo.
df.filter(like='lo', axis = 1) ## valor ## 0 5 ## 1 12 ## 2 8 ## 3 30Filtro de dados
Indexação booleana
É possível fazer o filtro de dados com um índice booleano
df[[False, True, False, True]] ## nome valor ## 1 A2 12 ## 3 A4 30A indexação pode ser feita de forma automática, com uma comparação lógica
df[df['valor'] > 10] ## nome valor ## 1 A2 12 ## 3 A4 30
Criando expressões mais complexas
df[(df['valor'] >= 5) & (df['valor'] < 12)] ## nome valor ## 0 A1 5 ## 2 A3 8 As mesmas consultas podem ser realizadas com o método query().
df.query('valor >= 5 & valor < 12') ## nome valor ## 0 A1 5 ## 2 A3 8Filtro de dados
Dica: Filtragem e substituição (where)
df_2 = df.copy()df_2[df_2['valor'] > 10] = -99; print(df_2)## nome valor## 0 A1 5## 1 -99 -99## 2 A3 8## 3 -99 -99Uma forma mais elegante, trabalhando com where
df.where(df['valor'] < 10, -99)## nome valor## 0 A1 5## 1 -99 -99## 2 A3 8## 3 -99 -99Agrupamento e agregação
Agrupar e agregar dados ajuda na análise dos dados. É através deste processo que técnicas de análise exploratória de dados e estatística descritiva podem ser aplicadas em diferentes grupos de dados.
Agrupamento e agregação
Agregações são operações aplicadas sobre os dados que resultam em um conjunto de valores
Algumas funções de agregação
- sum();
- max();
- min();
- mean().
Agrupamento e agregação
Agregando Series
s = pd.Series([1, 2, 3])Calculando o somatório
s.sum() ## 6Valor mínimo
s.min() ## 1Agregando DataFrames
df = pd.DataFrame([[1, 2, 3], [4, 5, 6]])Calculando o valor médio
df.mean() ## 0 2.5 ## 1 3.5 ## 2 4.5 ## dtype: float64Valor máximo
df.max() ## 0 4 ## 1 5 ## 2 6 ## dtype: int64Agrupamento e agregação
No processo de agrupamento, os dados são agrupados com base em suas características. Para tal finalidade, o Pandas disponibiliza o método groupby.
Agrupando dados
df = pd.DataFrame({ 'nome': ['Tel1', 'Tel2', 'Tel3'], 'tipo': ['antigo', 'novo', 'novo'] }) agrupado = df.groupby('tipo')Tipo retornado
print(agrupado) ## <pandas.core.groupby.generic.DataFrameGroupBy object at 0x7f02ba759ad0>Recuperando os grupos gerados
agrupado.groups ## {'antigo': Int64Index([0], dtype='int64'), 'novo': Int64Index([1, 2], dtype='int64')}Agrupamento + Agregação =
Agrupamento + Agregação
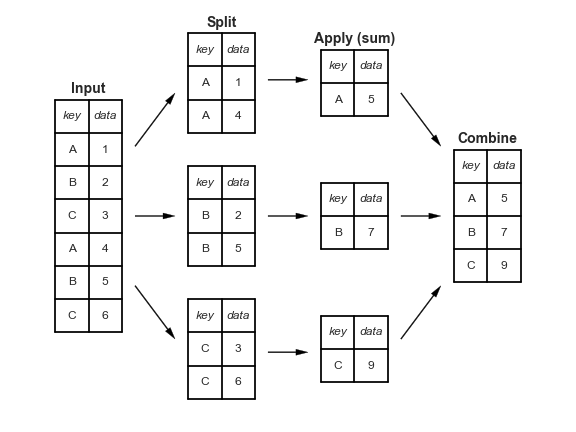
Agrupamento + Agregação
Exemplo de utilização de agrupamento com agregação
df = pd.DataFrame({ 'nome': ['ana', 'maria', 'felipe', 'joão'], 'idade': [19, 19, 20, 20], 'dinheiro': [150, 150, 100, 100] }) Agrupando pela idade e contando os elementos
# Esta é a etapa de divir, da definição do Hedley agrupado = df.groupby('idade') ## Esta é a etapa de aplicação e junçaõ, definida pelo Hedley print(agrupado.count()) ## nome dinheiro ## idade ## 19 2 2 ## 20 2 2Média de dinheiro por idade
agrupado['dinheiro'].mean() ## idade ## 19 150 ## 20 100 ## Name: dinheiro, dtype: int64A idade que recebe mais dinheiro
agrupado['dinheiro'].sum() ## idade ## 19 300 ## 20 200 ## Name: dinheiro, dtype: int64Leitura de dados
Leitura de dados
Com o Pandas é possível não só realizar o processamento dos dados, mas também a leitura e escrita destes. A biblioteca suporta diversos formatos, sendo alguns deles CSV, JSON e Excel.
Carregando arquivo CSV
data = pd.read_csv("data/titanic.csv", sep = ',') Os dados carregados convergem para Series e DataFrames.
type(data) ## <class 'pandas.core.frame.DataFrame'>Salvando os dados em CSV
data.to_csv('data/resultados.csv')Exemplo de análise de dados
Exemplo de análise de dados
Dados de 45 mil meteoritos que cairam na Terra, publicados pela NASA.
Carregando os dados
import pandas as pd data = pd.read_csv("data/meteorite-landings.csv") type(data) ## <class 'pandas.core.frame.DataFrame'>Verificando os atributos dos dados. A descrição de cada atributo está disponível na página dos dados
data.columns ## Index(['name', 'id', 'nametype', 'recclass', 'mass', 'fall', 'year', 'reclat', ## 'reclong', 'GeoLocation'], ## dtype='object')
Separando os dados por tipo de meteorito
df_valid = data[data['nametype'] == 'Valid'] df_relict = data[data['nametype'] == 'Relict']Verificando as quantidades de cada tipo
print("Valid: {} | Relict: {}".format( df_valid.shape[0], df_relict.shape[0] )) ## Valid: 45641 | Relict: 75Exemplo de análise de dados
Verificando a massa média de cada tipo de meteorito
df_groupby_nametype = data.groupby('nametype') df_groupby_nametype['mass'].mean() ## nametype ## Relict 0.121269 ## Valid 13285.656127 ## Name: mass, dtype: float64
Façamos a contagem dos tipos de meteoritos
df_groupedby_recclass = data.groupby('recclass') df_groupedby_recclass['recclass'].count().head(n = 3) ## recclass ## Acapulcoite 54 ## Acapulcoite/Lodranite 6 ## Acapulcoite/lodranite 3 ## Name: recclass, dtype: int64Por fim, façamos o filtro do conjunto de dados pela quantidade de massa
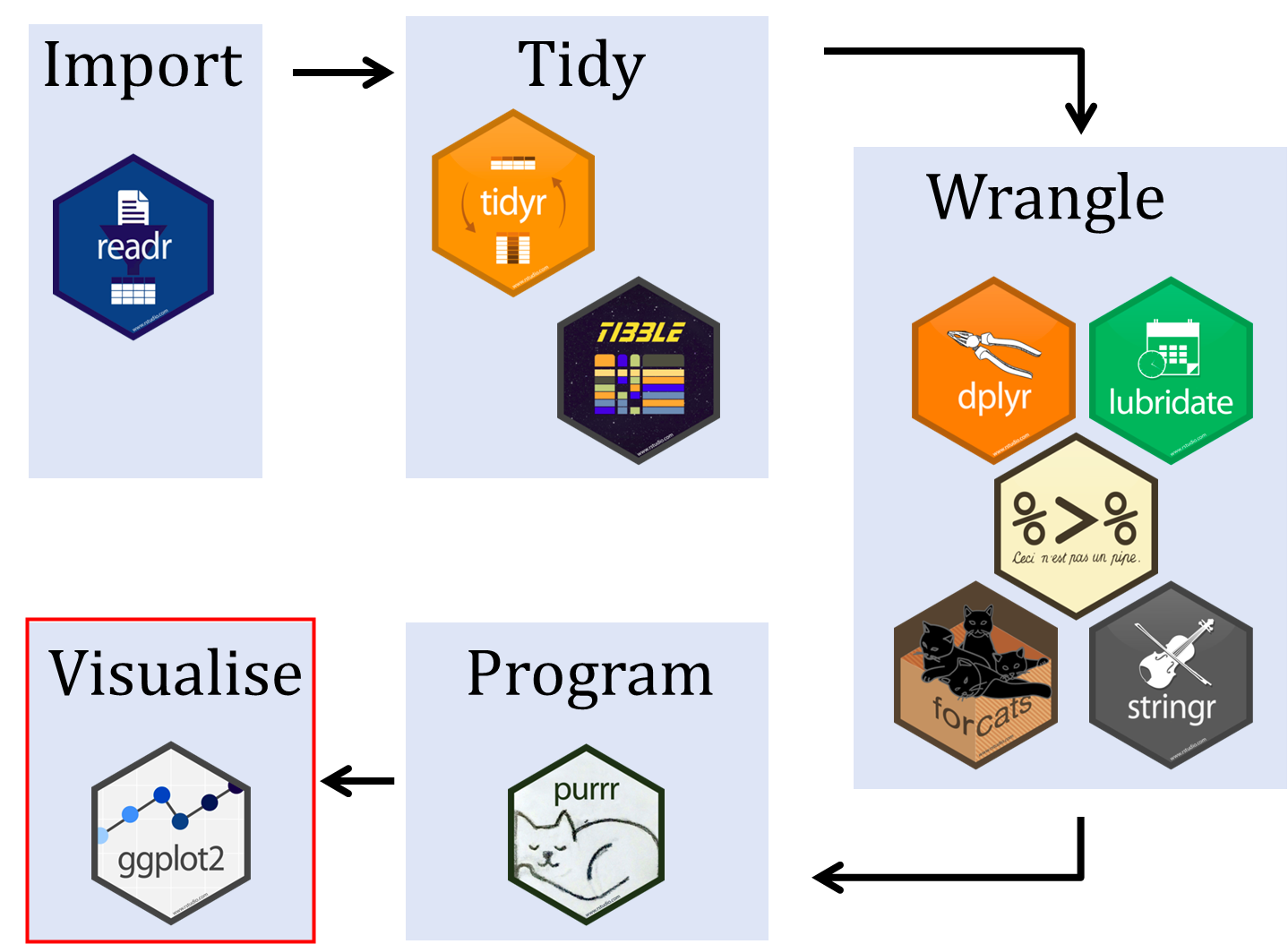
# Filtragem (Maior que 500 gramas) df_gt_mass_500 = data[data['mass'] > 500] df_gt_mass_500.shape # Quantidade bem menor ## (7036, 10)Visualização de dados
Pacotes de visualização
As bibliotecas de visualização de dados ggplot2 e plotnine são baseadas na obra The Grammar of Graphics, a qual apresenta uma grámatica para elaboração de gráficos. Tal gramática é composta por camadas, as quais descrevem os componentes do gráfico.
Camadas de componentes gráficos

Pacotes de visualização
As bibliotecas de visualização de dados ggplot2 e plotnine são baseadas na obra The Grammar of Graphics, a qual apresenta uma grámatica para elaboração de gráficos. Tal gramática é composta por camadas, as quais descrevem os componentes do gráfico.
Camadas de componentes gráficos

Sintaxe do ggplot/plotnine
ggplot(data = <DATA>, aes(<MAPPINGS>)) + <GEOM_FUNCTION>( mapping = aes(<MAPPINGS>), stat = <STAT>, position = <POSITION>) + <COORDINATE_FUNCTION> + <FACET_FUNCTION>Mapeamento estético
A estética descreve cada aspecto de um dado elemento gráfico. Descrevemos as posições (position) por um valor x e y, mas outros sistemas de coordenadas são possíveis. É possível alterar a forma (shape), tamanho (size) e cor (size) dos elementos.
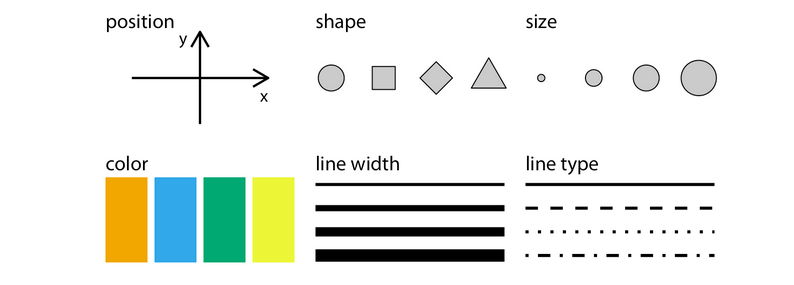 Fundamentals of Data Visualization - Claus O. Wilke
Fundamentals of Data Visualization - Claus O. Wilke
Objetos geométricos

Fundamentals of Data Visualization - Claus O. Wilke
Exemplo com R - Gráfico de dispersão
Mapeamento estético
library(ggplot2) ggplot(iris, aes(x = Petal.Width, y = Petal.Length, color = Species))
Exemplo com R - Gráfico de dispersão
Mapeamento estético
library(ggplot2) ggplot(iris, aes(x = Petal.Width, y = Petal.Length, color = Species))
Objeto geométrico
ggplot(iris, aes(x = Petal.Width, y = Petal.Length, color = Species)) + geom_point()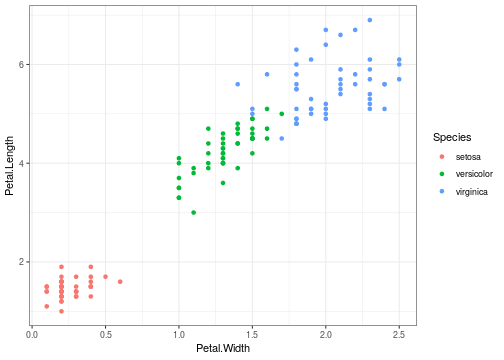
Exemplo com R - Gráfico de colunas
Mapeamento estético e objeto geométrico
ggplot(data = diamonds) + stat_count(mapping = aes(x = cut))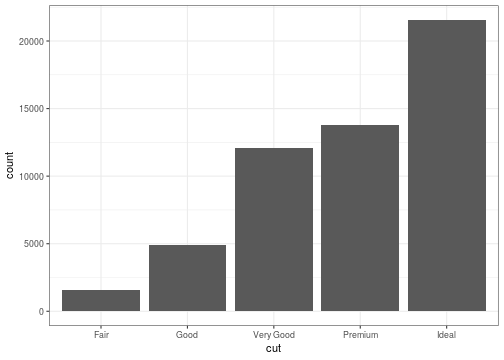
Exemplo com R - Gráfico de colunas
Mapeamento estético e objeto geométrico
ggplot(data = diamonds) + stat_count(mapping = aes(x = cut))
Sistema de coordenadas
ggplot(data = diamonds) + stat_count(mapping = aes(x = cut)) + coord_flip()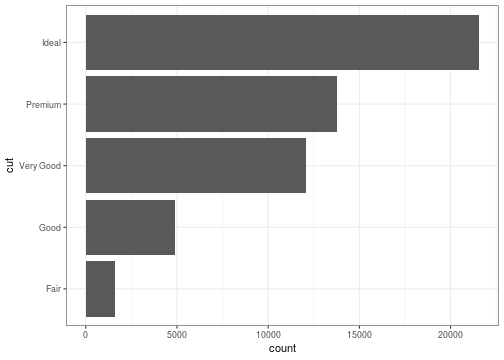
Exemplo com Python - Gráfico de dispersão
Mapeamento estético
from plotnine import *from plotnine.data import mtcars, diamondsggplot(mtcars, aes(x = 'mpg', y = 'disp'))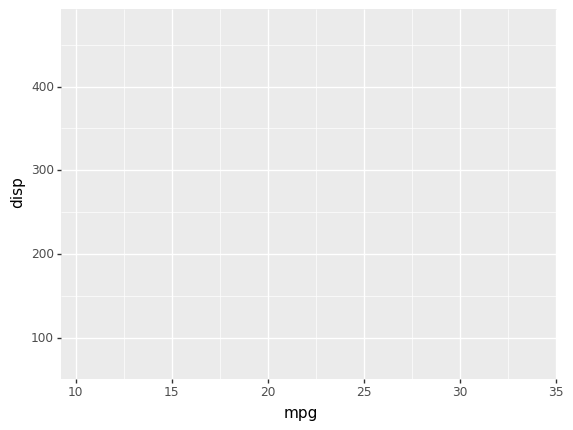
Exemplo com Python - Gráfico de dispersão
Mapeamento estético
from plotnine import *from plotnine.data import mtcars, diamondsggplot(mtcars, aes(x = 'mpg', y = 'disp'))
Objeto geométrico
(ggplot(mtcars, aes(x = 'mpg', y = 'disp', color = 'factor(am)')) + geom_point())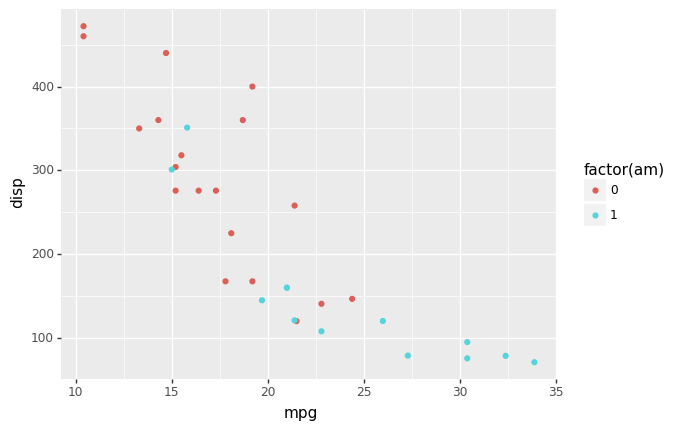
Exemplo com Python - Gráfico de colunas
Mapeamento estético e objeto geométrico
(ggplot(data = diamonds) + stat_count(mapping = aes(x = 'cut')))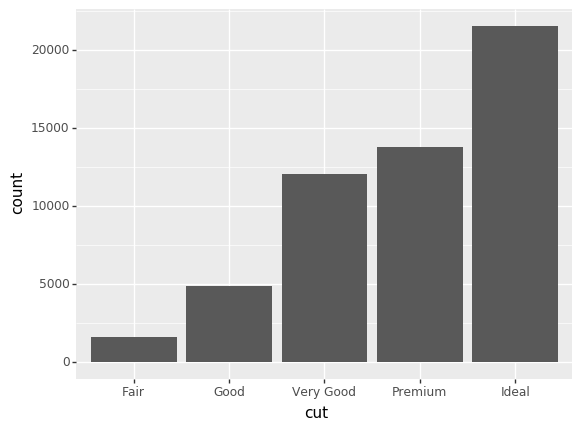
Exemplo com Python - Gráfico de colunas
Mapeamento estético e objeto geométrico
(ggplot(data = diamonds) + stat_count(mapping = aes(x = 'cut')))
Sistema de coordenadas
(ggplot(data = diamonds) + stat_count(mapping = aes(x = 'cut')) + coord_flip())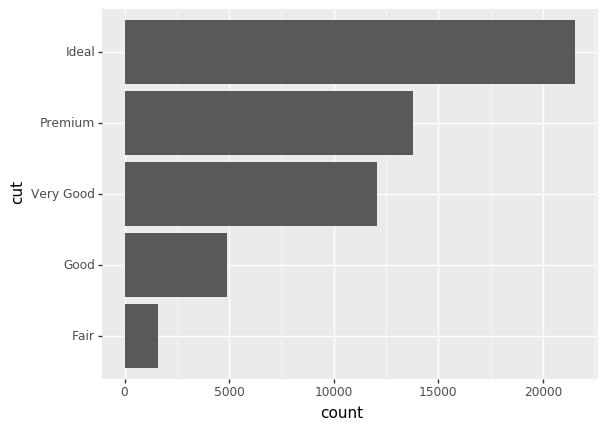
Indo além
Exemplo - Gráfico de coordenadas paralelas
iris %>% dplyr::mutate(id = 1:nrow(iris)) %>% tidyr::gather(atributos, valores, -Species, -id) %>% ggplot(., aes(x = atributos, y = valores, color = Species, group = id)) + geom_line(size=0.55) + labs(x = "Atributos", y = "Valores", title = "Coordenadas Paralelas - Iris", caption = "Fonte: dataAt") + theme_bw() + theme(plot.title = element_text(hjust= 0.5, margin = margin(b = 7)))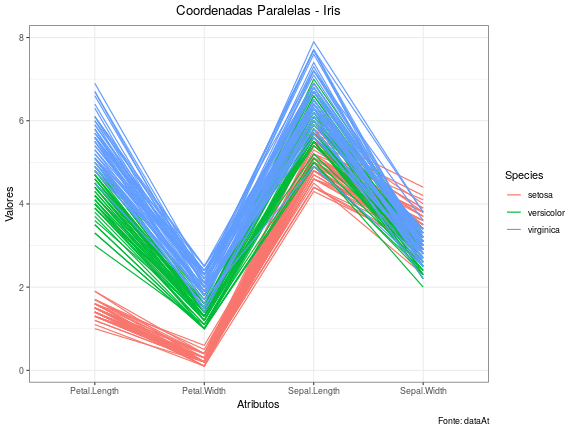
Exemplo - Gráfico de coordenadas paralelas
iris %>% dplyr::mutate(id = 1:nrow(iris)) %>% tidyr::gather(atributos, valores, -Species, -id) %>% ggplot(., aes(x = atributos, y = valores, color = Species, group = id)) + geom_line(size=0.55) + facet_grid(~Species) + labs(x = "Atributos", y = "Valores", title = "Coordenadas Paralelas - Iris") + theme_bw() + theme(plot.title = element_text(hjust= 0.5, margin = margin(b = 7)), axis.text.x = element_text(angle = 90))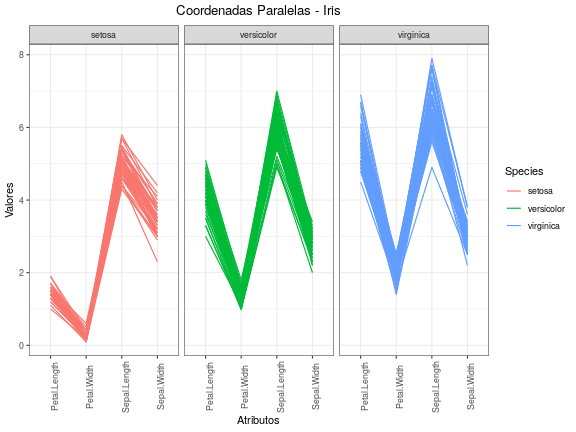
Extensões do ggplot
(Somente em R)
Lemon package
amost_diam %>% ggplot(., aes(x = as.factor(cut), y = price, color = clarity)) + geom_point(position=position_jitter(width=0.08)) + coord_flex_cart(bottom=brackets_horisontal(), left=capped_vertical('both')) + theme_light() + theme(panel.border=element_blank(), axis.line = element_line(), plot.title = element_text(hjust= 0.5, margin = margin(b = 7))) + labs(x = "Qualidade do corte", y = "Preço em US", title = "Gráfico de bolhas - Diamonds")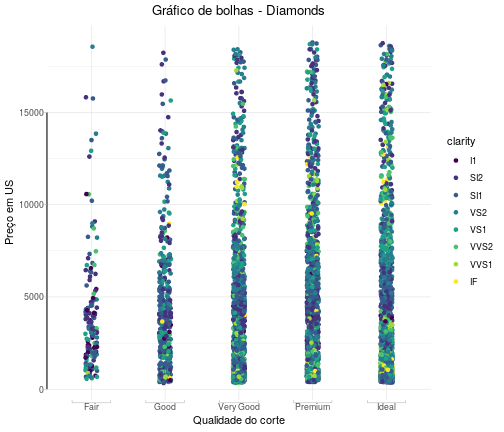
Obrigado!